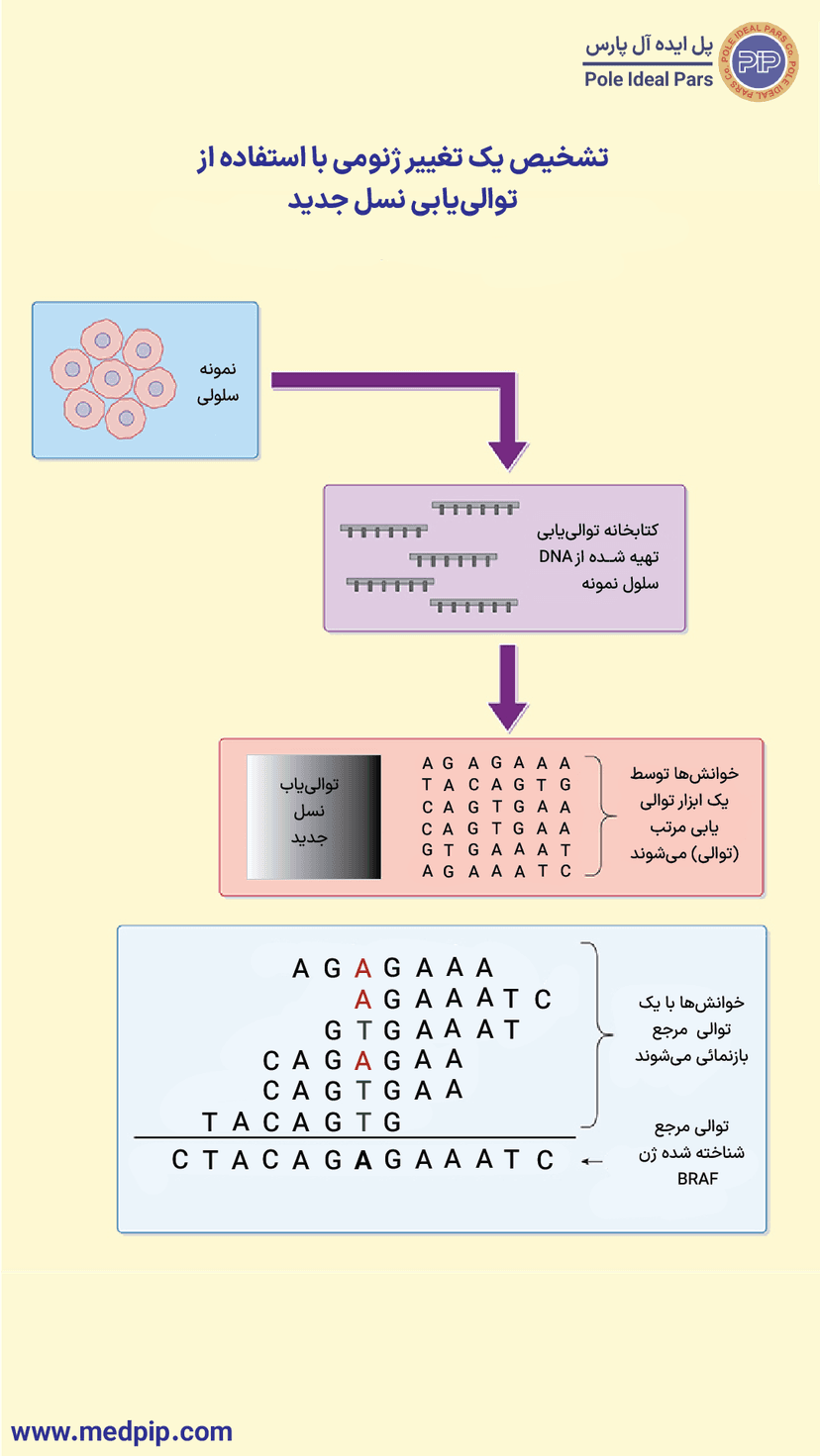
تشخیص یک تغییر ژنتیکی با استفاده از روش توالییابی نسل جدید
این بخش نحوه استفاده از توالییابی نسل جدید را برای تشخیص انواع تک نوکلئوتیدی در یک نمونه بیمار توصیف میکند. به طور معمول، این نوع از تشخیص تنوع با استفاده از توالی خوانش کوتاه انجام میشود. در نمونه بیمار که از بیوپسی تومور به دست آمده است و حاوی یک جهش هتروزیگوت منفرد در ژن BRAF به نام "BRAF V600E" است، تقریباً نیمی از DNA ایزوله شده دارای این نوع (واریانت) خواهد بود در حالی که نیمی دیگر این گونه را ندارند.
همانطور که در شکل نشان داده شده است، DNA از سلولهای بیمار جدا میشود. قطعه قطعه میشود. در یک ابزار توالییابی بارگذاری میشود و توالی "خواندهها" متشکل از بخشهای کوتاهی از نوکلئوتیدها تولید میشوند.
با استفاده از این دادهها، این خوانشها با یک توالی مرجع برای این ژن با تراز کردن خوانشها در جایی که پایگاههای آنها با مرجع مکمل هستند، همانطور که نشان داده شده است، مطابقت داده میشوند.
هنگام انجام این تراز، برای برخی از موقعیتها، پایگاههای تک تک به طور مداوم با توالی مرجع مطابقت ندارند، در حالی که سایر بازها در خوانشهای مربوطه خود تراز میشوند.
در پایین شکل نشان داده شده است که این برای نیمی از خوانشهای نگاشت شده صادق است. به عنوان مثال، در نیمی از خوانشها همخوانی A وجود دارد و برای نیمی دیگر، عدم همخوانی T وجود دارد.
باز ناهمساز با رنگ سبز برجسته شده است. نیمی از خوانشهای نگاشت شده که با مرجع برای موقعیت مورد نظر مطابقت دارند به رنگ قرمز مشخص شدهاند.
این نشان میدهد که نمونه برای جهش از A به T در این موقعیت در ژن BRAF هتروزیگوت است.